ماده اولیه: RNA
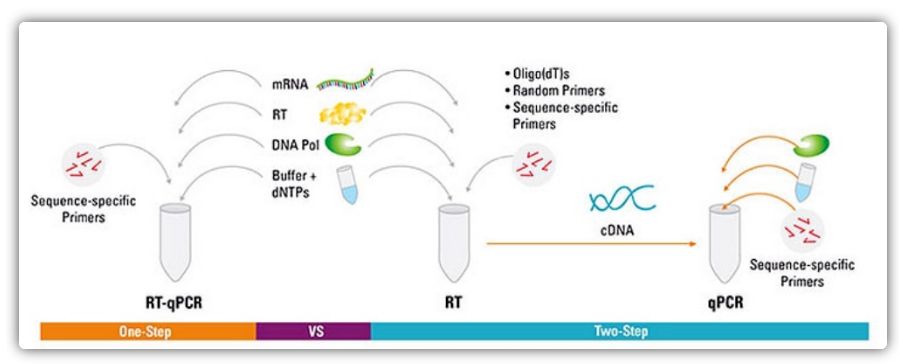
PCR رونویسی معکوس کمی (RT-qPCR) یک روش تجربی است که در آزمایشات PCR با استفاده از RNA به عنوان ماده اولیه استفاده می شود.در این روش ابتدا RNA تام یا RNA پیام رسان (mRNA) توسط ترانس کریپتاز معکوس به DNA مکمل (cDNA) رونویسی می شود.پس از آن، یک واکنش qPCR با استفاده از cDNA به عنوان یک الگو انجام شد.RT-qPCR در انواع کاربردهای زیست شناسی مولکولی، از جمله تجزیه و تحلیل بیان ژن، اعتبارسنجی تداخل RNA، اعتبارسنجی ریزآرایه، تشخیص پاتوژن، آزمایش ژنتیکی و تحقیقات بیماری استفاده شده است.
روش های یک مرحله ای و دو مرحله ای برای RT-qPCR
RT-qPCR را می توان با روش یک مرحله ای یا دو مرحله ای انجام داد.RT-qPCR یک مرحله ای رونویسی معکوس و تقویت PCR را ترکیب می کند و به رونویسی معکوس و DNA پلیمراز اجازه می دهد تا واکنش را در همان لوله تحت شرایط بافر یکسان تکمیل کنند.RT-qPCR یک مرحله ای فقط به استفاده از پرایمرهای توالی خاص نیاز دارد.در RT-qPCR دو مرحله ای، رونویسی معکوس و تقویت PCR در دو لوله، با استفاده از بافرهای بهینه شده مختلف، شرایط واکنش و استراتژی های طراحی پرایمر انجام می شود.
| مزیت - فایده - سود - منفعت | عیب | |
| یک قدم | این روش خطای تجربی کمتری دارد زیرا هر دو واکنش در یک لوله انجام می شود
مراحل کمتر پیپتینگ خطر آلودگی را کاهش می دهد
مناسب برای تقویت / غربالگری با توان بالا، سریع و قابل تکرار | واکنش های دو مرحله ای را نمی توان به طور جداگانه بهینه کرد
از آنجایی که شرایط واکنش با ترکیب واکنش دو مرحله ای به خطر می افتد، حساسیت به خوبی روش دو مرحله ای نیست.
تعداد اهداف شناسایی شده توسط یک نمونه کوچک است |
| دو قدم | توانایی ایجاد کتابخانه های cDNA پایدار که می توانند برای مدت زمان طولانی ذخیره شوند و در واکنش های متعدد استفاده شوند.
ژنهای هدف و ژنهای مرجع را میتوان از یک کتابخانه cDNA بدون نیاز به چندین کتابخانه cDNA تکثیر کرد.
بافرهای واکنش و شرایط واکنشی که بهینه سازی تک واکنش ها را امکان پذیر می کند
انتخاب انعطاف پذیر شرایط ماشه | استفاده از لوله های متعدد و مراحل بیشتر پیپت کردن، خطر آلودگی DNA را افزایش می دهد. و زمان بر است.
نسبت به روش تک مرحله ای به بهینه سازی بیشتری نیاز دارد |
محصولات مرتبط:
RT-qPCR Easyᵀᴹ (یک مرحله ای)-SYBR Green I
RT-qPCR Easyᵀᴹ (یک مرحله) - Taqman
RT Easyᵀᴹ I Master Premix برای سنتز CDNA رشته اول
کیت Real Time PCR Easyᵀᴹ-SYBR Green I
انتخاب RNA تام و mRNA
هنگام طراحی آزمایش RT-qPCR، تصمیم گیری در مورد استفاده از RNA تام یا mRNA خالص به عنوان الگویی برای رونویسی معکوس مهم است.اگرچه mRNA ممکن است بتواند حساسیت کمی بالاتری ایجاد کند، RNA کل هنوز هم اغلب استفاده می شود.دلیل این امر این است که RNA کل دارای مزیت مهم تری به عنوان ماده اولیه نسبت به mRNA است.اول، این فرآیند به مراحل تصفیه کمتری نیاز دارد، که بازیابی کمی بهتر الگو و عادی سازی بهتر نتایج را برای شروع شماره سلول تضمین می کند.دوم، از مرحله غنیسازی mRNA جلوگیری میکند، که میتواند از احتمال نتایج کج به دلیل بازیابی متفاوت mRNAهای مختلف جلوگیری کند.به طور کلی، از آنجایی که در بیشتر کاربردها، کمیت نسبی ژن هدف مهمتر از حساسیت مطلق تشخیص است، RNA کل در بیشتر موارد مناسب تر است.
آغازگر رونویسی معکوس
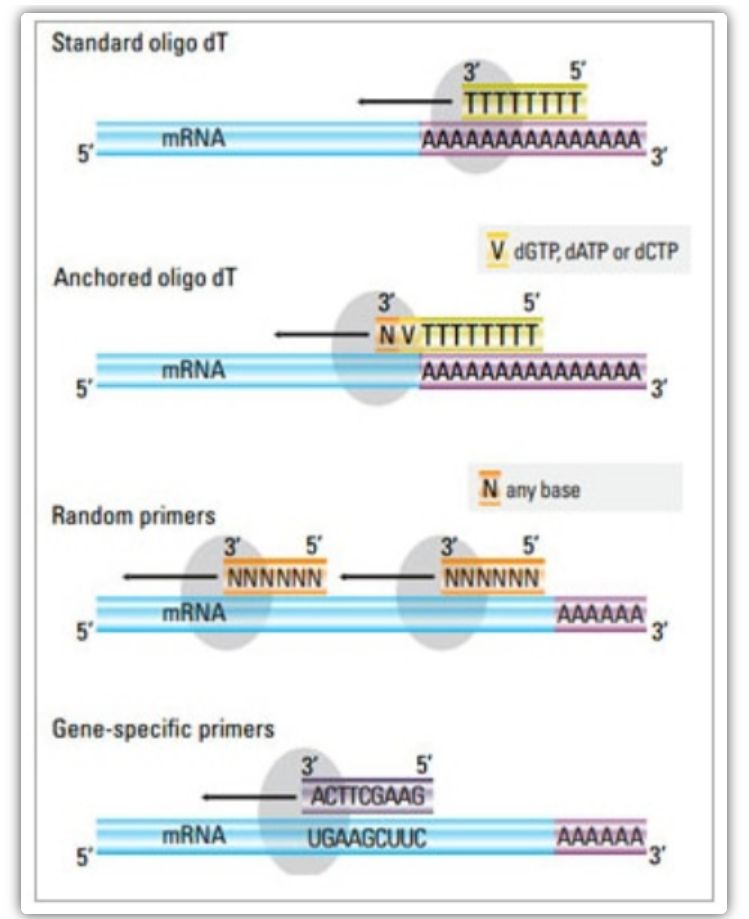
در روش دو مرحله ای، از سه روش مختلف می توان برای پرایم واکنش cDNA استفاده کرد: پرایمرهای oligo(dT)، پرایمرهای تصادفی یا پرایمرهای اختصاصی توالی.به طور معمول، پرایمرهای oligo(dT) و پرایمرهای تصادفی به صورت ترکیبی استفاده می شوند.این آغازگرها به رشته mRNA الگو بازپخت می شوند و ترانس کریپتاز معکوس را با نقطه شروع سنتز فراهم می کنند.
| انتخاب پرایمر | ساختار و عملکرد | مزیت - فایده - سود - منفعت | عیب |
| پرایمر الیگو(dT) (یا پرایمر لنگر الیگو(dT)) | بازپخت گسترده به باقی مانده های تیمین در دم پلی (A) mRNA.پرایمر لنگر الیگو(dT) حاوی یک G، C یا A در انتهای 3' (محل لنگر) است. | سنتز cDNA تمام قد از mRNA دم پلی (A).
زمانی که مواد اولیه کمتری در دسترس باشد قابل استفاده است
محل اتصال تضمین می کند که پرایمر الیگو(dT) به دم پلی(A) 5' mRNA متصل می شود. | فقط برای تقویت ژن با دم پلی (A) مناسب است
cDNA کوتاه شده را از محل پرایم*2 در poly(A) بدست آورید
جهت اتصال به انتهای 3'*
*در صورت استفاده از پرایمرهای لنگر الیگو(dT) این احتمال به حداقل می رسد |
| پرایمر تصادفی
| 6 تا 9 باز طول دارد که می تواند در طول رونویسی RNA به چندین مکان بازپخت | آنیل به همه RNA ها (tRNA، rRNA و mRNA)
مناسب برای رونوشت هایی با ساختار ثانویه قابل توجه، یا زمانی که مواد اولیه کمتری در دسترس است
بازده cDNA بالا | cDNA از تمام RNA رونویسی معکوس می شود، که معمولا مطلوب نیست و ممکن است سیگنال mRNA هدف را رقیق کند.
cDNA کوتاه شده را دریافت کنید |
| آغازگرهای توالی خاص | آغازگرهای سفارشی که توالی های mRNA خاصی را هدف قرار می دهند | کتابخانه cDNA خاص
بهبود حساسیت
استفاده از پرایمرهای qPCR معکوس | فقط به سنتز یک ژن هدف محدود می شود |
ترانس کریپتاز معکوس
ترانس کریپتاز معکوس آنزیمی است که از RNA برای سنتز DNA استفاده می کند.برخی از ترانس کریپتازهای معکوس دارای فعالیت RNase هستند و می توانند رشته های RNA را در رشته های هیبریدی RNA-DNA پس از رونویسی تجزیه کنند.اگر فعالیت آنزیمی RNase نداشته باشد، RNaseH می تواند برای کارایی qPCR بالاتر اضافه شود.آنزیم های رایج مورد استفاده شامل رونوشت معکوس ویروس لوسمی موشی مولونی و رونوشت معکوس ویروس میلوبلاستوم پرندگان است.برای RT-qPCR، انتخاب یک ترانس کریپتاز معکوس با پایداری حرارتی بالاتر ایده آل است، به طوری که سنتز cDNA را بتوان در دماهای بالاتر انجام داد و از رونویسی موفقیت آمیز RNA ها با ساختار ثانویه بالاتر اطمینان حاصل کرد، در حالی که فعالیت کامل آنها در طول واکنش حفظ می شود و در نتیجه بازده cDNA بالاتری ایجاد می شود.
محصولات مرتبط:
Foreasy M-MLV Reverse Transcriptase
فعالیت RNase H ترانس کریپتاز معکوس
RNaseH قادر است رشته های RNA را از دوبلکس های RNA-DNA تجزیه کند و امکان سنتز موثر DNA دو رشته ای را فراهم کند.با این حال، هنگام استفاده از mRNA طولانی به عنوان یک الگو، RNA ممکن است زودتر از موعد تجزیه شود و منجر به cDNA کوتاه شود.بنابراین، اگر سنتز رونوشت های طولانی مورد نظر باشد، اغلب مفید است که فعالیت RNaseH در طول شبیه سازی cDNA به حداقل برسد.در مقابل، رونوشتهای معکوس با فعالیت RNase H اغلب برای کاربردهای qPCR مفید هستند زیرا ذوب دوبلکسهای RNA-DNA را در طول اولین چرخه PCR افزایش میدهند.
طراحی پرایمر
پرایمرهای PCR مورد استفاده برای مرحله qPCR در RT-qPCR باید به طور ایدهآل طراحی شوند تا یک اتصال اگزون-اگزون را پوشش دهند، جایی که یک پرایمر تقویت به طور بالقوه میتواند یک مرز واقعی اگزون-اینترون را در بر بگیرد.از آنجایی که توالیهای DNA ژنومی حاوی اینترون تقویت نمیشوند، این طرح خطر مثبت کاذب تکثیر شده از DNA ژنومی آلوده را کاهش میدهد.
اگر پرایمرها را نتوان برای جداسازی اگزون ها یا مرزهای اگزون-اگزون طراحی کرد، ممکن است لازم باشد نمونه های RNA با DNase I یا dsDNase بدون RNase درمان شود تا آلودگی DNA ژنومی حذف شود.
کنترل RT-qPCR
یک کنترل منفی رونویسی معکوس (کنترل -RT) باید در تمام آزمایشهای RT-qPCR برای تشخیص آلودگی DNA (مانند DNA ژنومی یا محصولات PCR از واکنشهای قبلی) گنجانده شود.این کنترل شامل تمام اجزای واکنش به جز ترانس کریپتاز معکوس است.از آنجایی که رونویسی معکوس با این کنترل اتفاق نمی افتد، در صورت مشاهده تقویت PCR، احتمال آلودگی از DNA بسیار زیاد است.
زمان ارسال: آگوست-02-2022








