1. جداسازی RNA با کیفیت بالا:
سنتز موفق cDNA از RNA با کیفیت بالا حاصل می شود.RNA با کیفیت بالا حداقل باید تمام طول و عاری از مهارکننده های ترانس کریپتاز معکوس مانند EDTA یا SDS باشد.کیفیت RNA تعیین کننده حداکثر مقدار اطلاعات توالی است که می توانید در cDNA رونویسی کنید.یک روش متداول خالص سازی RNA یک روش یک مرحله ای با استفاده از گوانیدین ایزوتیوسیانات/فنل اسیدی است.برای جلوگیری از آلودگی توسط مقادیر کمی از RNase، RNA جدا شده از نمونه های غنی از RNase (مانند پانکراس) باید در فرمالدئید ذخیره شود تا RNA با کیفیت بالا، به ویژه برای ذخیره سازی طولانی مدت حفظ شود.RNA استخراج شده از کبد موش اساساً پس از نگهداری در آب به مدت یک هفته تجزیه شد، در حالی که RNA استخراج شده از طحال موش پس از نگهداری در آب به مدت 3 سال پایدار ماند.علاوه بر این، رونوشتهای طولانیتر از 4 کیلوبایت نسبت به رونوشتهای کوچک به تخریب توسط RNaseهای ردیابی حساستر هستند.برای افزایش پایداری نمونه های RNA ذخیره شده، RNA را می توان در فرمامید دیونیزه حل کرد و در دمای 70- درجه سانتی گراد نگهداری کرد.فرمامید مورد استفاده برای حفظ RNA باید عاری از بقایای تجزیه کننده RNA باشد.RNA لوزالمعده را می توان حداقل برای یک سال در فرمامید حفظ کرد.هنگام آماده شدن برای استفاده از RNA، می توانید از روش زیر برای رسوب RNA استفاده کنید: NaCl را به 0.2M و 4 برابر حجم اتانول اضافه کنید، به مدت 3-5 دقیقه در دمای اتاق قرار دهید و به مدت 5 دقیقه در 10000×g سانتریفیوژ کنید.
2. از رونوشت معکوس RNaseH-غیر فعال (RNaseH-) استفاده کنید:
مهارکنندههای RNase اغلب به واکنشهای رونویسی معکوس اضافه میشوند تا طول و بازده سنتز cDNA را افزایش دهند.مهارکنندههای RNase باید در طی واکنش سنتز رشته اول در حضور بافر و یک عامل کاهنده (مانند DTT) اضافه شوند، زیرا فرآیند قبل از سنتز cDNA باعث دناتوره شدن بازدارنده میشود و در نتیجه RNase متصل را آزاد میکند که میتواند RNA را تجزیه کند.مهارکننده های پروتئین RNase فقط از تجزیه RNA توسط RNase A, B, C جلوگیری می کنند و مانع RNase روی پوست نمی شوند، بنابراین مراقب باشید با وجود استفاده از این مهارکننده ها، RNase را از انگشتان خود وارد نکنید.
ترانس کریپتاز معکوس تبدیل RNA به cDNA را کاتالیز می کند.هر دو M-MLV و AMV علاوه بر فعالیت پلیمراز خود دارای فعالیت RNaseH درون زا هستند.فعالیت RNaseH و فعالیت پلیمراز برای رشته هیبریدی تشکیل شده بین الگوی RNA و پرایمر DNA یا رشته گسترش cDNA با یکدیگر رقابت می کنند و رشته RNA را در مجموعه RNA:DNA تجزیه می کنند.الگوی RNA تجزیه شده توسط فعالیت RNaseH دیگر نمی تواند به عنوان یک بستر موثر برای سنتز cDNA عمل کند، که باعث کاهش بازده و طول سنتز cDNA می شود.بنابراین، حذف یا کاهش شدید فعالیت RNaseH ترانس کریپتاز معکوس مفید خواهد بود.
SuperScript Ⅱ ترانس کریپتاز معکوس، RNaseH- MMLV رونوشت معکوس و ترمواسکریپت رونوشت معکوس، RNaseH- AMV، می توانند مقدار بیشتر و cDNA تمام طول بیشتری نسبت به MMLV و AMV به دست آورند.حساسیت RT-PCR تحت تأثیر میزان سنتز cDNA قرار خواهد گرفت.ThermoScript بسیار حساس تر از AMV است.اندازه محصولات RT-PCR به دلیل توانایی ترانس کریپتاز معکوس برای سنتز cDNA محدود می شود، به ویژه هنگام شبیه سازی cDNA های بزرگتر.در مقایسه با MMLV، SuperScripⅡ به طور قابل توجهی بازده محصولات طولانی RT-PCR را افزایش داد.ترانس کریپتاز معکوس RNaseH همچنین پایداری حرارتی را افزایش داده است، بنابراین واکنش را می توان در دماهای بالاتر از دمای معمولی 37-42 درجه سانتی گراد انجام داد.تحت شرایط سنتز پیشنهادی، از پرایمر oligo(dT) و 10 μCi [α-P]dCTP استفاده کنید.بازده کل رشته اول با استفاده از روش بارش TCA محاسبه شد.cDNA تمام طول با استفاده از باندهای مرتب شده با اندازه جدا شده و بر روی ژل آگارز قلیایی شمارش شد.
3. دمای انکوباسیون را برای رونویسی معکوس افزایش دهید:
دمای انکوباسیون بالاتر به باز کردن ساختار ثانویه RNA کمک می کند و بازده واکنش را افزایش می دهد.برای اکثر الگوهای RNA، انکوباسیون RNA و پرایمرها در دمای 65 درجه سانتیگراد بدون بافر یا نمک، و به دنبال آن سرد شدن سریع روی یخ، اکثر ساختارهای ثانویه را از بین می برد و به پرایمرها اجازه اتصال می دهد.با این حال، برخی از الگوها حتی پس از دناتوره شدن حرارتی، هنوز ساختارهای ثانویه دارند.تقویت این قالب های دشوار را می توان با استفاده از ThermoScript Reverse Transcriptase و قرار دادن واکنش رونویسی معکوس در دمای بالاتر برای بهبود تقویت انجام داد.دمای انکوباسیون بالاتر نیز می تواند ویژگی را افزایش دهد، به ویژه هنگامی که پرایمرهای اختصاصی ژن (GSP) برای سنتز cDNA استفاده می شود (به فصل 3 مراجعه کنید).در صورت استفاده از GSP، اطمینان حاصل کنید که Tm پرایمرها با دمای انکوباسیون مورد انتظار برابر است.از پرایمرهای oligo(dT) و تصادفی بالای 60 درجه سانتی گراد استفاده نکنید.پرایمرهای تصادفی نیاز به انکوباسیون در دمای 25 درجه سانتیگراد به مدت 10 دقیقه قبل از افزایش تا 60 درجه سانتیگراد دارند.علاوه بر استفاده از دمای رونویسی معکوس بالاتر، ویژگی را می توان با انتقال مستقیم مخلوط RNA/پرایمر از دمای دناتوراسیون 65 درجه سانتی گراد به دمای جوجه کشی رونویسی معکوس و افزودن مخلوط واکنش 2× از پیش گرم شده (سنتز شروع داغ cDNA) بهبود بخشید.این رویکرد به جلوگیری از جفت شدن پایه های بین مولکولی که در دماهای پایین تر رخ می دهد کمک می کند.تغییر دمای چندگانه مورد نیاز برای RT-PCR را می توان با استفاده از یک سیکلر حرارتی ساده کرد.
پلیمراز مقاوم در برابر حرارت Tth به عنوان یک DNA پلیمراز در حضور Mg2+ و به عنوان یک RNA پلیمراز در حضور Mn2+ عمل می کند.می توان آن را در دمای حداکثر 65 درجه سانتی گراد گرم نگه داشت.با این حال، حضور Mn2+ در طی PCR، وفاداری را کاهش میدهد، که باعث میشود Tth پلیمراز برای تقویت با دقت بالا، مانند شبیهسازی cDNA، مناسب نباشد.علاوه بر این، Tth دارای راندمان رونویسی معکوس پایینی است که حساسیت را کاهش می دهد، و از آنجایی که رونویسی معکوس و PCR را می توان با یک آنزیم انجام داد، واکنش های کنترلی بدون رونویسی معکوس را نمی توان برای مقایسه محصولات تقویت cDNA با DNA ژنومی آلوده استفاده کرد.محصولات تقویتی جدا شدند.
4. افزودنی هایی که رونویسی معکوس را ترویج می کنند:
افزودنی هایی از جمله گلیسرول و DMSO به واکنش سنتز رشته اول اضافه می شود که می تواند پایداری اسید نوکلئیک دو رشته ای را کاهش دهد و ساختار ثانویه RNA را باز کند.تا 20٪ گلیسرول یا 10٪ DMSO را می توان بدون تأثیر بر فعالیت SuperScript II یا MMLV اضافه کرد.AMV همچنین می تواند تا 20٪ گلیسرول را بدون از دست دادن فعالیت تحمل کند.به منظور به حداکثر رساندن حساسیت RT-PCR در واکنش رونویسی معکوس SuperScriptⅡ، گلیسرول 10% را می توان اضافه کرد و در دمای 45 درجه سانتی گراد انکوبه کرد.اگر 10/1 محصول واکنش رونویسی معکوس به PCR اضافه شود، غلظت گلیسرول در واکنش تقویتی 0.4 درصد است که برای مهار PCR کافی نیست.
5. درمان RNaseH:
درمان واکنش های سنتز cDNA با RNaseH قبل از PCR می تواند حساسیت را افزایش دهد.برای برخی از الگوها، تصور می شود که RNA در واکنش سنتز cDNA از اتصال محصولات تقویتی جلوگیری می کند، در این صورت درمان RNaseH می تواند حساسیت را افزایش دهد.به طور کلی، درمان RNaseH هنگام تقویت الگوهای هدف cDNA تمام طول طولانی تر، مانند اسکروز توبروس II با کپی کم، ضروری است.برای این الگوی دشوار، درمان RNaseH سیگنال تولید شده توسط SuperScript II یا cDNA سنتز شده با AMV را افزایش داد.برای اکثر واکنشهای RT-PCR، درمان RNaseH اختیاری است، زیرا مرحله دناتوراسیون PCR در دمای 95 درجه سانتیگراد به طور کلی RNA را در مجموعه RNA:DNA هیدرولیز میکند.
6. بهبود روش تشخیص RNA کوچک:
RT-PCR به ویژه زمانی چالش برانگیز است که فقط مقادیر کمی از RNA در دسترس باشد.گلیکوژن اضافه شده به عنوان یک حامل در طول جداسازی RNA به افزایش بازده نمونه های کوچک کمک می کند.گلیکوژن بدون RNase را می توان همزمان با افزودن Trizol اضافه کرد.گلیکوژن محلول در آب است و می توان آن را در فاز آبی با RNA نگه داشت تا به رسوب بعدی کمک کند.برای نمونه های کمتر از 50 میلی گرم بافت یا 106 سلول کشت شده، غلظت توصیه شده گلیکوژن بدون RNase 250 میکروگرم بر میلی لیتر است.
افزودن BSA استیله به واکنش رونویسی معکوس با استفاده از SuperScript II میتواند حساسیت را افزایش دهد و برای مقادیر کم RNA، کاهش مقدار SuperScript II و افزودن 40 واحد بازدارنده نوکلئاز RNaseOut میتواند سطح تشخیص را افزایش دهد.اگر از گلیکوژن در فرآیند جداسازی RNA استفاده می شود، همچنان توصیه می شود هنگام استفاده از SuperScript II برای واکنش رونویسی معکوس، BSA یا RNase بازدارنده اضافه شود.
ویژگی RT-PCR را افزایش دهید
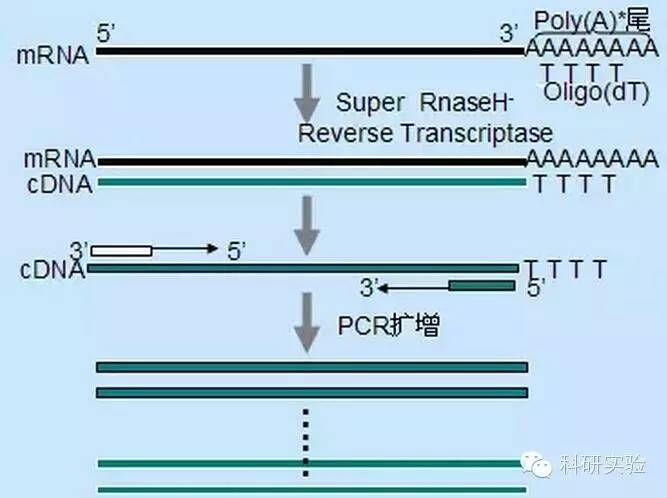
1. سنتز CND:
سنتز cDNA رشته اول را می توان با استفاده از سه روش مختلف آغاز کرد که ویژگی نسبی آنها بر مقدار و نوع cDNA سنتز شده تأثیر می گذارد.
روش پرایمر تصادفی کمترین ویژگی را در بین سه روش داشت.پرایمرها در چندین مکان در سراسر رونوشت بازپخت می شوند و cDNA های کوتاه و با طول جزئی تولید می کنند.این روش اغلب برای به دست آوردن توالی های انتهایی 5' و برای به دست آوردن cDNA از الگوهای RNA با نواحی ساختار ثانویه یا با مکان های پایانی که نمی توانند توسط ترانس کریپتاز معکوس تکرار شوند استفاده می شود.برای به دست آوردن طولانی ترین cDNA، نسبت پرایمرها به RNA در هر نمونه RNA باید به صورت تجربی تعیین شود.غلظت شروع پرایمرهای تصادفی از 50 تا 250 نانوگرم در هر 20 میکرولیتر واکنش متغیر بود.از آنجایی که cDNA سنتز شده از RNA کل با استفاده از پرایمرهای تصادفی در درجه اول RNA ریبوزومی است، پلی(A)+RNA به طور کلی به عنوان الگو انتخاب می شود.
پرایمرهای Oligo(dT) از پرایمرهای تصادفی اختصاصی تر هستند.با دم پلی (A) که در انتهای 3' اکثر mRNA های یوکاریوتی یافت می شود هیبرید می شود.از آنجایی که RNA پلی(A)+ تقریباً 1 تا 2 درصد RNA کل است، مقدار و پیچیدگی cDNA بسیار کمتر از پرایمرهای تصادفی است.به دلیل ویژگی بالای آن، الیگو(dT) معمولاً به بهینه سازی نسبت RNA به آغازگرها و انتخاب پلی (A)+ نیاز ندارد.استفاده از 0.5μg oligo(dT) در هر 20μl سیستم واکنش توصیه می شود.oligo(dT)12-18 برای اکثر RT-PCR مناسب است.سیستم ThermoScript RT-PCR به دلیل پایداری حرارتی بهتر برای دماهای جوجه کشی بالاتر، oligo(dT)20 را ارائه می دهد.
پرایمرهای اختصاصی ژن (GSP) اختصاصی ترین آغازگرها برای مرحله رونویسی معکوس هستند.GSP یک الیگونوکلئوتید آنتی سنس است که می تواند به طور خاص با توالی هدف RNA هیبرید شود، برخلاف پرایمرهای تصادفی یا الیگو(dT)، که به همه RNA ها بازپخت می شوند.قوانین مشابهی که برای طراحی پرایمرهای PCR استفاده می شود، در طراحی GSP در واکنش های رونویسی معکوس نیز اعمال می شود.GSP می تواند همان توالی پرایمر تقویتی باشد که به 3'ترین انتهای mRNA بازپخت می شود، یا GSP می تواند برای بازپخت پایین دست پرایمر تقویت معکوس طراحی شود.برای برخی از افراد تکثیر شده، بیش از یک پرایمر آنتی سنس باید برای موفقیت RT-PCR طراحی شود زیرا ساختار ثانویه RNA هدف ممکن است از اتصال پرایمر جلوگیری کند.توصیه می شود از 1 pmol GSP آنتی سنس در واکنش سنتز رشته اول 20 میکرولیتر استفاده شود.
2. دمای انکوباسیون را برای رونویسی معکوس افزایش دهید:
به منظور استفاده کامل از مزیت کامل ویژگی GSP، یک رونوشت معکوس با پایداری حرارتی بالاتر باید استفاده شود.ترانس کریپتازهای معکوس مقاوم در برابر حرارت را می توان در دماهای بالاتر برای افزایش سختی واکنش انکوبه کرد.به عنوان مثال، اگر یک GSP در دمای 55 درجه سانتیگراد بازپخت شود، اگر AMV یا M-MLV برای رونویسی معکوس در شدت کم 37 درجه سانتیگراد استفاده شود، از ویژگی GSP به طور کامل استفاده نمی شود.با این حال، SuperScript II و ThermoScript می توانند در دمای 50 درجه سانتیگراد یا بالاتر واکنش نشان دهند، که محصولات غیر اختصاصی تولید شده در دماهای پایین تر را حذف می کند.برای حداکثر ویژگی، مخلوط RNA/پرایمر را می توان مستقیماً از دمای دناتوراسیون 65 درجه سانتی گراد به دمای انکوباسیون رونویسی معکوس منتقل کرد و به مخلوط واکنش 2× از قبل گرم شده (شروع داغ سنتز cDNA) اضافه کرد.این به جلوگیری از جفت شدن پایه های بین مولکولی در دماهای پایین کمک می کند.انتقال دمای چندگانه مورد نیاز برای RT-PCR را می توان با استفاده از یک سیکلر حرارتی ساده کرد.
3. آلودگی DNA ژنومی را کاهش می دهد:
یک مشکل بالقوه ای که با RT-PCR مواجه می شود، آلودگی DNA ژنومی در RNA است.استفاده از یک روش جداسازی RNA خوب، مانند معرف تریزول، میزان DNA ژنومی آلوده کننده آماده سازی RNA را کاهش می دهد.برای جلوگیری از محصولات مشتق شده از DNA ژنومی، RNA را می توان با DNase I درجه تقویت کرد تا DNA آلوده را قبل از رونویسی معکوس حذف کرد.هضم DNase I با انکوبه کردن نمونه ها در 2.0 میلی مولار EDTA به مدت 10 دقیقه در دمای 65 درجه سانتیگراد خاتمه یافت.EDTA می تواند یون های منیزیم را کلات کند و از هیدرولیز RNA وابسته به یون منیزیم در دماهای بالا جلوگیری کند.
به منظور جداسازی cDNA تکثیر شده از محصولات تکثیر DNA ژنومی آلوده، پرایمرهایی را می توان طراحی کرد که هر کدام برای جداسازی اگزون ها بازپخت شوند.محصولات PCR مشتق شده از cDNA کوتاه تر از محصولات حاصل از DNA ژنومی آلوده هستند.علاوه بر این، یک آزمایش کنترل بدون رونویسی معکوس بر روی هر الگوی RNA انجام شد تا مشخص شود آیا یک قطعه داده شده از DNA ژنومی یا cDNA مشتق شده است.محصول PCR به دست آمده بدون رونویسی معکوس از ژنوم مشتق شده است.
زمان ارسال: مه-16-2023