1. دانش پایه (اگر می خواهید قسمت آزمایشی را ببینید، لطفا مستقیماً به قسمت دوم منتقل کنید)
به عنوان یک واکنش مشتق از PCR معمولی، Real Time PCR عمدتاً تغییر مقدار محصول تقویت را در هر چرخه واکنش تقویت PCR در زمان واقعی از طریق تغییر سیگنال فلورسانس نظارت میکند و الگوی شروع را از طریق رابطه بین مقدار ct و منحنی استاندارد تجزیه و تحلیل کمی میکند.
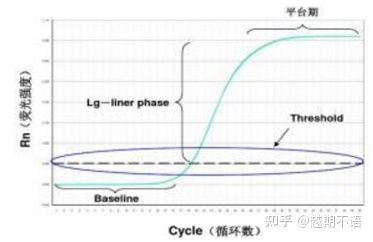
داده های خاص RT-PCR هستندخط پایه, آستانه فلورسانسومقدار Ct.
| خط مبنا: | مقدار فلورسانس سیکل 3-15 خط پایه (پایه) است که به دلیل خطای گاه به گاه اندازه گیری ایجاد می شود. |
| آستانه (آستانه): | اشاره به حد تشخیص فلورسانس تنظیم شده در یک موقعیت مناسب در ناحیه رشد نمایی منحنی تقویت، معمولاً 10 برابر انحراف استاندارد خط پایه است. |
| مقدار CT: | تعداد چرخه های PCR زمانی است که مقدار فلورسانس در هر لوله واکنش به آستانه می رسد. مقدار Ct با مقدار قالب اولیه نسبت معکوس دارد. |
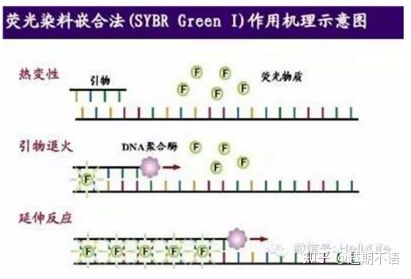
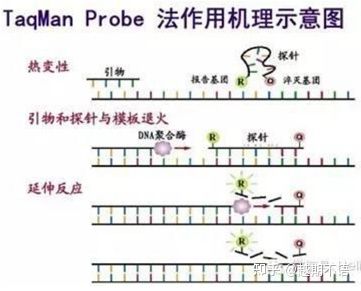
روش های رایج برچسب زدن برای RT-PCR:
| روش | مزیت - فایده - سود - منفعت | کمبود | دامنه کاربرد |
| SYBR GreenⅠ | کاربرد گسترده، حساس، ارزان و راحت | نیاز پرایمر زیاد است، مستعد باندهای غیر اختصاصی است | این برای تجزیه و تحلیل کمی ژن های مختلف هدف، تحقیق در مورد بیان ژن، و تحقیق در مورد حیوانات و گیاهان نوترکیب تراریخته مناسب است. |
| TaqMan | ویژگی خوب و تکرارپذیری بالا | قیمت بالا است و فقط برای اهداف خاص مناسب است. | تشخیص پاتوژن، تحقیقات ژن مقاومت دارویی، ارزیابی اثربخشی دارو، تشخیص بیماری های ژنتیکی. |
| چراغ مولکولی | ویژگی بالا، فلورسانس، پس زمینه کم | قیمت بالا است، فقط برای یک هدف خاص مناسب است، طراحی دشوار است و قیمت بالا است. | تجزیه و تحلیل ژن خاص، تجزیه و تحلیل SNP |
2. مراحل آزمایشی
2.1 درباره گروه بندی تجربی- باید چندین چاه در گروه وجود داشته باشد و باید تکرارهای بیولوژیکی وجود داشته باشد.
| ① | کنترل خالی | برای تشخیص وضعیت رشد سلولی در آزمایشات استفاده می شود |
| ② | siRNA کنترل منفی (توالی siRNA غیر اختصاصی) | ویژگی عمل RNAi را نشان دهید.siRNA ممکن است پاسخ استرس غیر اختصاصی را در غلظت 200 نانومولار القا کند. |
| ③ | کنترل معرف ترانسفکشن | حذف سمیت معرف ترانسفکشن برای سلول ها یا تأثیر بر بیان ژن هدف |
| ④ | siRNA در برابر ژن هدف | بیان ژن هدف را از بین ببرید |
| ⑤ (اختیاری) | siRNA مثبت | برای عیب یابی سیستم آزمایشی و مشکلات عملیاتی استفاده می شود |
| ⑥ (اختیاری) | siRNA کنترل فلورسنت | کارایی انتقال سلولی را می توان با میکروسکوپ مشاهده کرد |
2.2 اصول طراحی پرایمر
| اندازه قطعه تقویت شده | ترجیحا در 100-150bp |
| طول پرایمر | 18-25 جفت باز |
| محتوای GC | 30٪ - 70٪، ترجیحا 45 - 55٪ |
| مقدار Tm | 58-60 ℃ |
| توالی | اجتناب از T/C مداوم.A/G پیوسته |
| 3 دنباله پایانی | از GC rich یا AT rich اجتناب کنید.پایه ترمینال ترجیحاً G یا C است.بهتر است از T اجتناب کنید |
| مکمل بودن | از توالی های تکمیلی بیش از 3 باز در پرایمر یا بین دو پرایمر خودداری کنید |
| اختصاصی | برای تأیید ویژگی پرایمر از جستجوی انفجار استفاده کنید |
①SiRNA برای گونه خاص است و توالی گونه های مختلف متفاوت خواهد بود.
②SiRNA در پودر خشک شده یخ بسته بندی شده است که می تواند به مدت 2-4 هفته در دمای اتاق به طور پایدار نگهداری شود.
2.3 ابزارها یا معرف هایی که باید از قبل آماده شوند
| پرایمر (مرجع داخلی) | از جمله جلو و عقب دو |
| پرایمرها (ژن هدف) | از جمله جلو و عقب دو |
| RNA هدف Si (3 نوار) | به طور کلی، این شرکت 3 نوار را سنتز می کند و سپس یکی از سه نوار را با RT-PCR انتخاب می کند. |
| کیت ترانسفکشن | Lipo2000 و غیره |
| کیت استخراج سریع RNA | برای استخراج RNA پس از ترانسفکشن |
| کیت رونویسی معکوس سریع | برای سنتز cDNA |
| کیت تقویت PCR | 2×Super SYBR سبز qPCR Master Mix |
2.4 با توجه به مواردی که در مراحل آزمایشی خاص باید به آنها توجه شود:
① فرآیند انتقال siRNA
1. برای آبکاری، می توانید صفحه 24 چاهی، صفحه 12 چاهی یا صفحه 6 چاهی را انتخاب کنید (متوسط غلظت RNA پیشنهادی در هر چاه یک صفحه 24 چاهی حدود 100-300 نانوگرم در لیتر است) و تراکم ترانسفکشن بهینه سلول ها تا 60٪ -80٪ یا بیشتر است.
2. مراحل ترانسفکشن و الزامات خاص کاملاً مطابق با دستورالعمل است.
3. پس از ترانسفکشن، نمونه ها را می توان در عرض 24-72 ساعت برای تشخیص mRNA (RT-PCR) یا تشخیص پروتئین در 48-96 ساعت (WB) جمع آوری کرد.
② فرآیند استخراج RNA
1. جلوگیری از آلودگی توسط آنزیم های اگزوژن.این عمدتا شامل پوشیدن ماسک و دستکش به شدت است.با استفاده از نوک پیپت های استریل شده و لوله های EP.آب مورد استفاده در آزمایش باید بدون RNase باشد.
2. توصیه می شود دو بار به عنوان پیشنهاد شده در کیت استخراج سریع انجام شود، که واقعا خلوص و عملکرد را بهبود می بخشد.
3. مایع زباله نباید ستون RNA را لمس کند.
③ کمی RNA
پس از استخراج RNA، می توان آن را مستقیماً با Nanodrop اندازه گیری کرد و حداقل قرائت می تواند تا 10ng/ul کم باشد.
④فرایند رونویسی معکوس
1. با توجه به حساسیت بالای RT-qPCR، حداقل باید 3 چاه موازی برای هر نمونه ساخته شود تا Ct بعدی بیش از حد متفاوت نباشد یا SD برای تجزیه و تحلیل آماری خیلی بزرگ نباشد.
2. مخلوط مستر را به طور مکرر منجمد و ذوب نکنید.
3. هر لوله / سوراخ باید با یک نوک جدید جایگزین شود!به طور مداوم از نوک پیپت یکسان برای افزودن نمونه استفاده نکنید!
4. فیلم متصل به صفحه 96 چاهی پس از افزودن نمونه باید با صفحه صاف شود.بهتر است قبل از قرار دادن آن روی دستگاه سانتریفیوژ کنید تا مایع روی دیواره لوله به سمت پایین جریان یابد و حباب های هوا را از بین ببرد.
⑤تحلیل منحنی مشترک
| بدون دوره رشد لگاریتمی | احتمالا غلظت بالای قالب |
| بدون مقدار CT | مراحل نادرست برای تشخیص سیگنال های فلورسنت؛ تخریب پرایمرها یا پروب ها - یکپارچگی آن را می توان با الکتروفورز PAGE تشخیص داد. مقدار ناکافی الگو؛ تخریب قالب ها - اجتناب از ورود ناخالصی ها و انجماد و ذوب مکرر در آماده سازی نمونه. |
| Ct> 38 | راندمان تقویت پایین؛محصول PCR خیلی طولانی است.اجزای مختلف واکنش تجزیه می شوند |
| منحنی تقویت خطی | کاوشگرها ممکن است با چرخه های مکرر انجماد و ذوب یا قرار گرفتن طولانی مدت در معرض نور تا حدی تخریب شوند. |
| تفاوت در سوراخ های تکراری به ویژه زیاد است | محلول واکنش کاملا ذوب نشده یا محلول واکنش مخلوط نشده است.حمام حرارتی دستگاه PCR توسط مواد فلورسنت آلوده شده است |
2.5 درباره تجزیه و تحلیل داده ها
تجزیه و تحلیل داده های qPCR را می توان به کمیت نسبی و کمیت مطلق تقسیم کرد.به عنوان مثال، سلول های گروه درمان در مقایسه با سلول های گروه کنترل،
چند بار mRNA ژن X تغییر می کند، این کمیت نسبی است.در تعداد معینی از سلول ها، mRNA ژن X
چند نسخه وجود دارد، این کمیت مطلق است.معمولاً آنچه در آزمایشگاه بیشتر استفاده می کنیم روش کمی نسبی است.معمولا،روش 2-ΔΔctبیشتر در آزمایشات استفاده می شود، بنابراین فقط این روش در اینجا به تفصیل معرفی می شود.
روش 2-ΔΔct: نتیجه به دست آمده تفاوت بیان ژن هدف در گروه آزمایش نسبت به ژن هدف در گروه کنترل است.لازم است که راندمان تکثیر هم ژن هدف و هم ژن مرجع داخلی نزدیک به 100 درصد باشد و انحراف نسبی نباید از 5 درصد تجاوز کند.
روش محاسبه به شرح زیر است:
گروه کنترل Δct = مقدار ct ژن هدف در گروه کنترل – مقدار ct ژن مرجع داخلی در گروه کنترل
Δct گروه آزمایش = مقدار ct ژن هدف در گروه آزمایش – مقدار ct ژن مرجع داخلی در گروه آزمایش
ΔΔct=Δct گروه آزمایش-Δct گروه کنترل
در نهایت، مضرب تفاوت در سطح بیان را محاسبه کنید:
تغییر Fold=2-ΔΔct (مطابق با تابع اکسل POWER است)
محصولات مرتبط:
زمان ارسال: مه-20-2023