ویژگی تشخیص
در بیشتر موارد، هدف از طراحی پرایمر به حداکثر رساندن ویژگی PCR است.این با تأثیر کم و بیش قابل پیش بینی بسیاری از متغیرها تعیین می شود.یکی از متغیرهای مهم توالی در انتهای 3 پرایمر است.
نکته مهم این است که سنجشهای PCR که برای اختصاصی بودن طراحی شدهاند، احتمال بیشتری دارد که راندمان بالا را در یک محدوده دینامیکی گسترده حفظ کنند، زیرا این روش محصولات تقویتی غیر اختصاصی تولید نمیکند، در نتیجه با معرفهای PCR رقابت میکند یا واکنش اصلی تقویت را مهار میکند.
البته، در برخی موارد، ویژگی مهم ترین نیست، به عنوان مثال، زمانی که هدف تعیین کمیت پاتوژن های نزدیک اما متفاوت است، طراحی، بهینه سازی و استانداردهای راستی آزمایی خاص مورد نیاز است.
منحنی ذوب یک روش استاندارد برای ارزیابی ویژگی آمپلیکون ها، حداقل از نظر تقویت یک هدف واحد است.با این حال، باید تأکید کرد که منحنیهای ذوب میتوانند گمراهکننده باشند، زیرا، برای مثال، میتوانند تحت تأثیر اثرات ترکیبی آغازگرهای غیربهینه و غلظتهای پایین قالب قرار بگیرند.
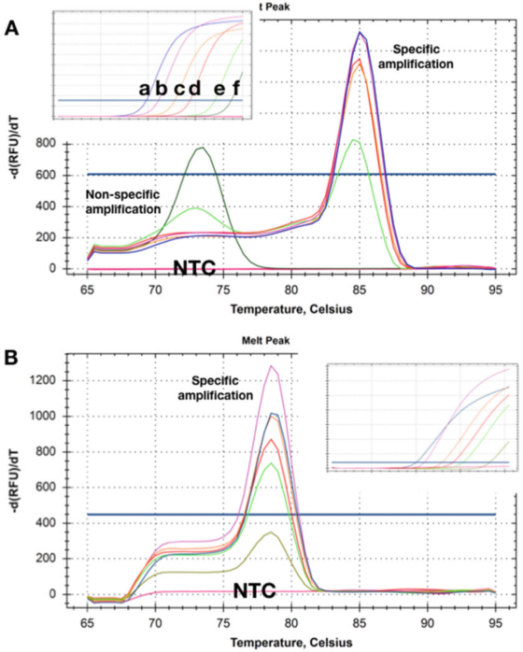
P5 |منحنی ذوب، جابجایی های Tm حاصل از دو تشخیص مقادیر مختلف دو DNA هدف را نشان می دهد.
الف. در غلظت های بالاتر (ad))، پس از اتمام اندازه گیری qPCR، هیچ دایمر پرایمر آشکاری وجود ندارد.با کاهش غلظت الگو به 50 نسخه (e)، یک محصول غیر اختصاصی ظاهر می شود و تنها محصول در کمترین غلظت (f) می شود.
ب. آزمون Tms یکسان را در تمام غلظت های هدف ثبت کرد و هیچ دایمر پرایمر آشکاری حتی در کمترین غلظت (5 نسخه) وجود نداشت.هنگام استفاده از این دو روش تشخیص، هیچ محصول تقویتی در NTC ها شناسایی نشد.
P5 منحنی های انحلال به دست آمده با نمونه هایی را نشان می دهد که در آنها الگو در غلظت های مختلف وجود دارد.P5a نشان می دهد که در دو پایین ترین غلظت، Tms محصولات تقویت غیر اختصاصی تولید شده کمتر از آمپلیکون های خاص است.
بدیهی است که این روش تشخیص نمی تواند به طور قابل اعتماد برای شناسایی اهدافی که در غلظت های پایین وجود دارند استفاده شود.
جالب اینجاست که NTCها، یعنی نمونههایی که اصلاً DNA ندارند، محصولات تکثیر (غیر اختصاصی) را ثبت نکردند، که نشان میدهد DNA ژنومی پسزمینه میتواند در تقویت/پلیمریزاسیون غیر اختصاصی شرکت کند.
گاهی اوقات چنین آغازگرهای پس زمینه و تقویت غیر اختصاصی قابل اصلاح نیستند، اما اغلب می توان یک روش تشخیص طراحی کرد که در هیچ غلظت قالب و NTC تقویت غیر اختصاصی نداشته باشد (P 5b).
در اینجا، حتی ثبت تقویت غلظت هدف با Cq 35، منحنی انحلال خاصی تولید می کند.به طور مشابه، NTC ها هیچ نشانه ای از تقویت غیر اختصاصی نشان ندادند.گاهی اوقات، رفتار تشخیص ممکن است وابسته به لیکور مادر باشد و تنها تقویت غیر اختصاصی در ترکیبات بافر خاصی که ممکن است مربوط به غلظت های مختلف Mg2+ باشد، تشخیص داده می شود.
پایداری تشخیص
بهینه سازی Ta یک مرحله مفید در تأیید تجربی و فرآیند بهینه سازی تشخیص qPCR است.با نشان دادن دما (یا محدوده دمایی) که کمترین Cq را بدون تقویت NTC ایجاد می کند، نشان مستقیمی از استحکام مجموعه پرایمر ارائه می دهد.
تفاوت دو تا چهار برابری در حساسیت ممکن است برای افرادی که بیان mRNA بالایی دارند مهم نباشد، اما برای تست های تشخیصی ممکن است به معنای تفاوت بین نتایج مثبت و منفی کاذب باشد.
خواص Ta پرایمرهای qPCR ممکن است بسیار متفاوت باشد.برخی از آزمایش ها چندان قوی نیستند و اگر تحت مقدار بهینه Ta پرایمرها انجام نشوند، به سرعت فرو می ریزند.
این مهم است زیرا این نوع تشخیص اغلب در دنیای واقعی مشکل ساز است و خلوص نمونه، غلظت DNA یا وجود DNA دیگر ممکن است بهینه نباشد.
علاوه بر این، تعداد کپی هدف ممکن است در محدوده وسیعی متفاوت باشد و معرفها، ظروف پلاستیکی یا ابزار ممکن است با مواردی که هنگام تنظیم آزمایش استفاده میشوند متفاوت باشند.
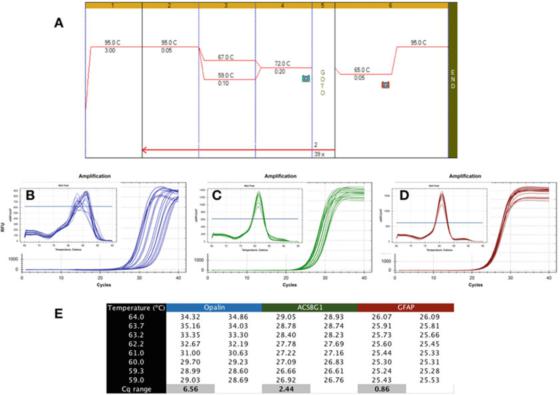
P6| گرادیان دما استحکام متفاوت تشخیص PCR را نشان می دهد.
الف. از Mastermix Sensifast SYBR Bioline (شماره کاتالوگ BIO-98050) برای انجام PCR روی cDNA تهیه شده از RNA مغز انسان استفاده کنید.
ب. از ابزار Bio-Rad's CFX qPCR برای ثبت نقشه تقویت و منحنی انحلال آپالن استفاده کنید (NM_033207، F: GCCATGGAGGAAAGTGACAGACC، R: CTCATGTGTGGGTGATCTCCTAGG).
ج. نمودار تقویت و منحنی ذوب ACSBG1 (NM_015162.4، F: CTACACTTCCGGCACCACTGG، R: GTCCACGTGATATTGTCTTGACTCAG).
D. نمودار تقویت و منحنی انحلال GFAP (NM_002055.5، F: TGGAGAGGAAGATTGAGTCGCTGG، R: CGAACCTCCTCTCTCGTGGATCTTC).
E. Cqs ثبت شده در دماهای بازپخت متفاوت، نشان دهنده تفاوت در Cq ثبت شده در یک گرادیان دمایی 7C است.
P6 یک نتیجه معمولی از یک آزمایش نامطلوب را نشان می دهد، که در آن qPCR با استفاده از گرادیان Tas بین 59C و 67C (P6a)، با استفاده از پرایمرهای سه ژن خاص مغز انسان انجام شد.
از نمودار تقویت مشاهده می شود که پرایمرهای Opalin بسیار دور از ایده آل هستند، زیرا محدوده Ta بهینه آنها بسیار باریک است (شکل 6b)، یعنی Cqs به طور گسترده پراکنده می شوند، در نتیجه Cqs به طور قابل توجهی با Cqs پایین بهینه آنها مقایسه می شود.
این روش تشخیص ناپایدار است و ممکن است منجر به تقویت نابهینه شود.بنابراین، این جفت پرایمر باید دوباره طراحی شود.علاوه بر این، تجزیه و تحلیل منحنی ذوب (inset) نشان می دهد که ویژگی این روش تشخیص نیز ممکن است مشکل ساز باشد، زیرا منحنی ذوب هر Ta متفاوت است.
روش تشخیص ACSBG1 نشان داده شده در P 6c قوی تر از روش تشخیص Opalin در بالا است، اما هنوز با ایده آل فاصله زیادی دارد و احتمالاً می توان آن را بهبود بخشید.
با این حال، ما تأکید می کنیم که هیچ ارتباط ضروری بین استحکام و ویژگی وجود ندارد، زیرا منحنی انحلال تولید شده توسط این روش تشخیص، مقدار پیک یکسانی را در تمام Tas (inset) نشان می دهد.
از سوی دیگر، تست استحکام بسیار متحملتر است و Cqs مشابهی را در طیف وسیعی از Tas تولید میکند، همانطور که در تست GFAP نشان داده شده در P 6d.
اختلاف Cqs بدست آمده در همان محدوده 8 درجه سانتیگراد کمتر از 1 است و منحنی انحلال (inset) مشخصه های تشخیص را در این محدوده دما تایید می کند.شایان ذکر است که Tas محاسبه شده و محدوده Ta واقعی ممکن است بسیار متفاوت باشد.
دستورالعمل های زیادی برای کمک به محققین در طراحی پرایمرهای کارآمد طراحی شده است که اکثر آنها بر اساس قوانینی از دیرباز تثبیت شده اند و توجه زیادی به پایان 3 پرایمرها شده است.اغلب توصیه می شود که یک G یا C در انتهای 3' و دو پایه G یا C (گیره GC) قرار دهید، اما از 5 پایه آخر بیش از دو پایه نباشد.
در عمل، این قوانین می تواند محققان را راهنمایی کند، اما لزوماً در هر شرایطی صحیح نیستند.
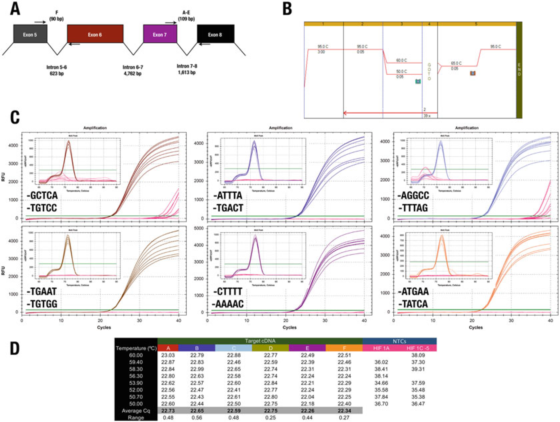
P7 |انتهای 3 پرایمر تأثیر کمی بر ویژگی یا کارایی دارد.
الف. موقعیت پرایمرهای ژن HIF-1α (NM_181054.2) انسانی.
ب. از مشروب مادری Agilent Brilliant III SYBR Green (Cat. No. 600882) برای تقویت شش مورد آزمایشی استفاده کنید.
ج. نمودار تقویت و منحنی ذوب توسط ابزار CFX qPCR Bio-Rad و پرایمرهای 3'end ثبت شده است.NTC ها با رنگ قرمز نشان داده شده اند.
D. رکورد Cqs هر آیتم آزمایشی
به عنوان مثال، نتیجه در P 7 با قانون پایان 3 در تضاد است.همه طرحها اساساً نتایج یکسانی را تولید میکنند و تنها دو ترکیب آغازگر منجر به تقویت غیر اختصاصی در NTC میشوند.
با این حال، ما نمی توانیم اثر کلیپ GC را پشتیبانی کنیم، زیرا در این حالت، استفاده از A یا T به عنوان حداکثر 30 پایه، ویژگی را کاهش نمی دهد.
تست C، جایی که پرایمر F به GGCC ختم میشود، Cqs را در NTCها ثبت کرد، که نشان میدهد ممکن است بخواهید از این توالیها در انتهای 30 اجتناب کنید.ما تأکید می کنیم که تنها راه برای تعیین بهترین توالی 3' پایانی یک جفت پرایمر، ارزیابی تجربی برخی آغازگرهای کاندید است.
راندمان تقویت
نکته مهم، اگرچه تشخیص غیراختصاصی PCR هرگز نمیتواند خاص شود، با تغییر آنزیم، مشروب مادر، مواد افزودنی و شرایط چرخهای، راندمان تقویت را میتوان به روشهای مختلف تنظیم و به حداکثر رساند.
برای ارزیابی کارایی تشخیص PCR، بهتر است از رقت سریال 10 یا 5 برابر اسید نوکلئیک هدف، یعنی "روش منحنی استاندارد" استفاده شود.
اگر از آمپلیکون های PCR یا اهداف DNA مصنوعی برای تولید یک منحنی استاندارد استفاده شود، رقت های سریالی این اهداف باید با مقدار ثابتی از DNA زمینه (مانند DNA ژنومی) مخلوط شوند.
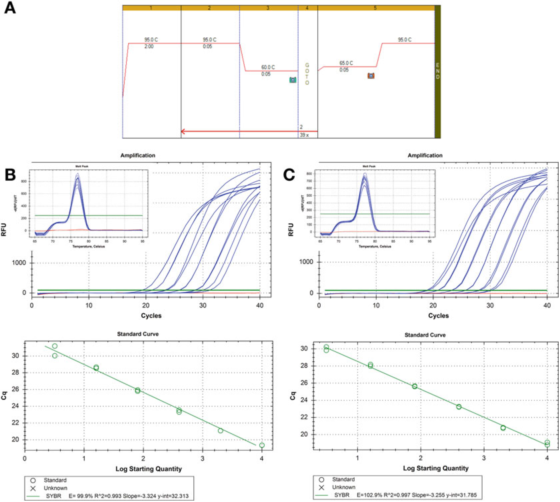
P8 |منحنی رقت برای ارزیابی کارایی PCR.
الف. از پرایمرها برای HIF-1 استفاده کنید: F: AAGAACTTTTAGGCCGCTCA و R: TGTCCTGTGGTGACTTGTCC و مستر میکس Agilent's Brilliant III SYBR Green (شماره کاتالوگ 600882) برای شرایط PCR و منحنی ذوب.
ب. 100 نانوگرم RNA رونویسی معکوس شد، 2 بار رقیق شد و نمونههای cDNA رقیقشده به صورت سریال 5 بار به 1 نانوگرم DNA ژنومی انسان رقیق شدند.منحنی ذوب در قسمت داخلی نشان داده شده است.
C. واکنش RT، رقتسازی و رقتسازی سریال برای نمونه cDNA دوم تکرار شد و نتایج مشابه بود.
P 8 دو منحنی استاندارد را نشان می دهد، با استفاده از روش تشخیص یکسان در دو نمونه cDNA مختلف، نتیجه کارایی یکسان است، حدود 100٪، و مقدار R2 نیز مشابه است، یعنی درجه تناسب بین داده های تجربی و خط رگرسیون یا درجه خطی بودن داده ها.
دو منحنی استاندارد قابل مقایسه هستند، اما دقیقاً یکسان نیستند.اگر هدف تعیین کمیت دقیق هدف باشد، باید توجه داشت که ارائه یک محاسبه عدد کپی بدون توضیح عدم قطعیت غیرقابل قبول است.
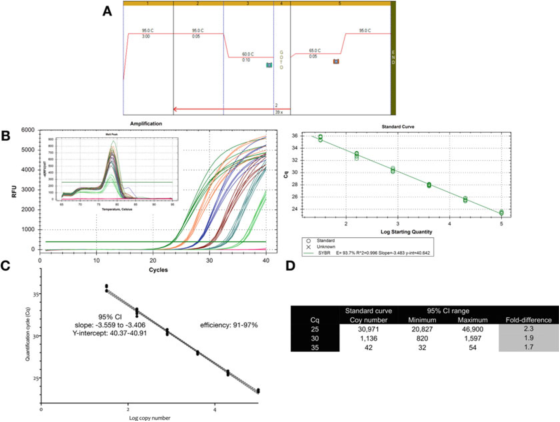
P9 |عدم قطعیت اندازه گیری مرتبط با کمی سازی با استفاده از یک منحنی استاندارد.
الف. از پرایمرهای GAPDH (NM_002046) برای انجام PCR و شرایط منحنی ذوب استفاده کنید.F: ACAGTTGCCATGTAGACC و R: TAACTGGTTGAGCACAGG و مستترمیکس Sensifast SYBR Bioline (شماره کاتالوگ BIO-98050).
ب. نمودار تقویت، منحنی ذوب و منحنی استاندارد ثبت شده با ابزار CFX qPCR Bio-Rad.
ج. نمودار منحنی استاندارد و فاصله اطمینان 95% (CI).
د- تعداد کپی و فاصله اطمینان 95% سه مقدار Cq حاصل از منحنی رقت.
P 9 نشان می دهد که برای یک آزمون بهینه شده، تغییرپذیری ذاتی یک منحنی استاندارد تقریباً 2 برابر است (95% فاصله اطمینان، حداقل تا حداکثر)، که ممکن است کوچکترین متغیری باشد که می توان انتظار داشت.
محصولات مرتبط:
زمان ارسال: سپتامبر 30-2021